jueves, 13 de abril de 2023
LAS "VACUNAS" COVID SUPERAN AMPLIAMENTE LA CANTIDAD ACEPTABLE DE ADN BACTERIANO (1ª PARTE)
Estudios recientes de Kevin McKernan, un destacado experto en métodos de secuenciación de ADN y ARN, han revelado que los lotes de las vacunas de ARNm modificado producidas tanto por Pfizer como por Moderna contienen una elevada proporción de ADN bacteriano contaminante. En total, el ADN representa hasta el 20-35% de los ácidos nucleicos contenidos en cada uno de los lotes de vacunas. Estas concentraciones alarmantemente elevadas superan con creces los niveles considerados seguros por organizaciones normativas como la Agencia Europea del Medicamento (EMA). Este documento resume las pruebas de esa contaminación por ADN y analiza qué posibles riesgos para la salud implica para los receptores de las vacunas.
1. El papel del ADN en la fabricación de vacunas de ARNm
1.1. Antecedentes generales
La mayoría de los lectores sabrán que
1.- los ARN sintéticos contenidos en las vacunas de ARNm COVID-19 codifican la proteína pico del SRAS-CoV-2;
2.- en las células vivas de los mamíferos, las instrucciones para construir una determinada molécula de proteína se almacenan como un gen dentro del ADN en el interior del núcleo;
3.- para construir una determinada molécula proteica, la célula transcribe primero su gen en ARN y modifica los dos extremos de esta molécula para formar ARN mensajero (ARNm). A continuación, el ARNm se transporta del núcleo al citoplasma, donde induce a las fábricas de proteínas de la célula -los ribosomas- a traducir la secuencia de nucleótidos del ARNm en la secuencia de aminoácidos correspondiente y ensamblar la proteína.
1.2. Pasos en la fabricación de vacunas de ARNm
Dado que la proteína de la espiga es una molécula grande, también lo es el ARNm que la codifica. La síntesis química total de grandes moléculas de ARNm no es práctica a escala. Por lo tanto, para obtener la molécula de ARNm que codifica la espiga, se imita in vitro el proceso por el cual las células producen sus propios ARNm. Esto implica los siguientes pasos:
1.- Se inserta una copia de ADN del gen de la proteína espiga en un plásmido bacteriano. Se trata de una molécula de ADN de doble cadena en forma de anillo que puede existir en una célula bacteriana independientemente del ADN cromosómico propio de la célula, y que también puede copiarse y transmitirse a ambas células hijas cuando la célula se divide.
2.- El plásmido recombinante (artificial) portador del gen de la proteína de la espiga se introduce en una célula de la especie bacteriana Escherichia coli (E. coli ). Dado que las células de E. coli se dividen muy rápidamente, esta única célula puede crecer en poco tiempo hasta convertirse en un gran número de células. Cada una de estas células progenitoras contendrá sus propias copias heredadas del plásmido y, por lo tanto, del gen de la proteína de la espiga. Aunque existe una cierta posibilidad de que el plásmido se pierda en algunas de las células progenitoras durante las sucesivas divisiones celulares, podemos reforzar su mantenimiento dándole un marcador seleccionable, que garantiza que sólo sobrevivirán las células que conserven el plásmido. En el caso de los plásmidos utilizados por Pfizer y Moderna, este marcador de selección es un gen que dota a las células huésped de resistencia al antibiótico kanamicina. Para aplicar la selección, basta con cultivar las bacterias en presencia de kanamicina.
3.- Después de cultivar un número suficiente de células bacterianas en un caldo nutritivo que contiene kanamicina, estas células se rompen y el ADN plasmídico se purifica de los otros componentes celulares bacterianos.
4.- Las moléculas de plásmido en forma de anillo se convierten a forma lineal utilizando una enzima de restricción, que corta ambas hebras de la molécula de ADN en un sitio específico y único que se encuentra aguas abajo del gen de la proteína espiga. Este paso es necesario para evitar la formación de moléculas de ARN demasiado largas, que podrían tener efectos indeseados in vivo. Las moléculas de ADN linealizadas pueden purificarse a partir de las circulares restantes, pero no se sabe cómo ni con qué eficacia puede hacerse esto en la producción de las vacunas de Pfizer y Moderna.
5.- Se utiliza una ARN polimerasa, en presencia de los nucleósidos y cofactores necesarios, para copiar el gen de la proteína de la espiga de la versión de ADN en el plásmido linealizado a la versión de ARNm. Tanto Pfizer como Moderna utilizan la ARN polimerasa T7, derivada del bacteriófago del mismo nombre. Esta enzima se une a una secuencia promotora también derivada de T7 que se ha introducido en el plásmido antes del gen de la proteína de la espiga. En esta fase, el nucleósido sintético N-metil-pseudouridina (mψU) se incorpora al ARN artificial en lugar del nucleósido natural uridina. Cuando se administra en forma de vacuna, el ARN modificado de esta manera es menos estimulante para el sistema inmunitario innato que el ARN que contiene la uridina natural. También se traduce más eficazmente en proteínas y, en determinadas condiciones, es más resistente a la degradación. Tanto las vacunas de ARNm de Pfizer como las de Moderna contienen mψU en lugar de uridina.
6.- Los dos extremos de cada molécula de ARN se acoplan enzimáticamente a ciertas moléculas que también se encuentran en estas posiciones dentro de los ARNm naturales de mamíferos, y que mejoran su actividad biológica y estabilidad in vivo.
Estos pasos proporcionan un ARNm funcional capaz de ordenar a los ribosomas de las células que produzcan la proteína de la espiga. Sin embargo, en esta fase el producto aún no es puro: todo el ADN molde de origen bacteriano sigue presente. Este último no debe incluirse en el producto farmacéutico final, ya que plantea riesgos para la salud de los receptores (véase la sección 4 en la próxima entrega de esta entrada). Para deshacerse de este ADN, se añade otra enzima llamada ADNasa. Esto debería romper el ADN en fragmentos más pequeños, que luego pueden separarse de las moléculas de ARN, mucho más grandes, mediante filtración y otras técnicas de purificación. En el último paso, el ARNm se combina con una mezcla de lípidos para empaquetarlo en nanopartículas lipídicas (LNP), que inducen a las células humanas a captar la molécula de ARNm y fabricar la proteína de la espiga.
2. ¿Qué sabíamos hasta ahora del problema de la contaminación por ADN?
En pocas palabras, muy poco. Los informes de evaluación de la FDA sobre ambas vacunas no mencionan el problema en absoluto. El informe de evaluación de la Agencia Europea del Medicamento (EMA) sobre la vacuna de Pfizer menciona que “La solidez de la etapa de digestión con DNasa no se considera ampliamente demostrada” [p. 17]. Un lenguaje similar se utiliza en el informe de la EMA sobre la vacuna Moderna [p. 19f]. Sin embargo, basándose únicamente en esta escasa información, es imposible decir si el problema se consideró grave y qué soluciones exigió el regulador, si es que exigió alguna.
3. Pruebas independientes sobre la contaminación por ADN de los productos de ARNm
Desde el 3 de abril de 2023, Kevin McKernan ha descrito sus recientes hallazgos en tres artículos en su sitio Substack. Los experimentos descritos en los dos primeros informes se llevaron a cabo con muestras de vacunas “bivalentes” recientemente introducidas por Pfizer y Moderna. Estas preparaciones se parecen a las anteriores “monovalentes” en su composición química, es decir, deben contener ARNm de gran pureza, envuelto con una mezcla de moléculas lipídicas (parecidas a las grasas) en nanopartículas de ARNm/lípido. La única diferencia entre las dos variedades es que las vacunas bivalentes contienen una mezcla de dos ARNm que codifican dos variantes antigénicas de la proteína de la espiga. Esto no tiene ninguna relación con el problema técnico de la contaminación por ADN como tal. Observamos, sin embargo, que el grado de contaminación por ADN puede variar entre los lotes de producción, y que hasta ahora sólo se ha caracterizado un pequeño número de lotes a este respecto.
3.1. El primer informe de McKernan
En un primer estudio, McKernan caracterizó tanto el ARN como el ADN contenidos en las vacunas de ARNm.
3.1.1. Extracción y caracterización directa de los ácidos nucleicos de las vacunas
El primer paso consistió en eliminar los lípidos para obtener los ácidos nucleicos puros. El método basado en disolventes que utilizó no discrimina entre ADN y ARN: si ambos están presentes, se recuperarán los dos. Los ácidos nucleicos extraídos se separaron por tamaños. Esto reveló no sólo las especies regulares esperadas de ARNm de espiga de longitud completa, sino también fragmentos más pequeños, que habían sido observados previamente tanto por los reguladores como en el trabajo publicado por uno de los fabricantes. Lo que es más sorprendente, también se encontraron especies de ARN más grandes que el ARNm de longitud completa. Estas especies siguen sin estar caracterizadas.
3.1.2. Amplificación de los ácidos nucleicos extraídos
Como paso preparatorio para determinar las secuencias nucleotídicas exactas de los ácidos nucleicos extraídos, se amplificaron mediante métodos de PCR. En el caso del ARN, la PCR fue precedida por la transcripción inversa en ADN utilizando una enzima específica (transcriptasa inversa). Dado que el objetivo principal de este estudio era estudiar el ARN y no el ADN, este paso de amplificación se sesgó en contra del ADN mediante la adición de actinomicina D, que en las condiciones experimentales dadas inhibe selectivamente la síntesis de ADN. En consecuencia, se recuperaron cantidades relativamente bajas de ADN en la muestra amplificada. Sin embargo, en el caso de la vacuna de Pfizer, la cantidad de ADN que se determinó que estaba presente ya superaba el límite arbitrariamente decidido por la EMA para la proporción máxima permisible de ADN por ARN.
3.1.3. Resultados de la secuenciación del ADN
Tanto con los productos de Pfizer como con los de Moderna, se obtuvieron secuencias de ADN de plásmidos de ADN completos, aunque seguía habiendo cierta ambigüedad en el caso de los plásmidos de Moderna. Por lo tanto, las características de las secuencias de plásmidos se discutirán en relación con el segundo estudio de McKernan, que utilizó ADN cada vez más puro para la secuenciación y, por lo tanto, proporcionó resultados más fiables.
3.2. El segundo informe de McKernan
El segundo estudio se centró en cuantificar y caracterizar la contaminación por ADN que se detectó cualitativamente en el primero.
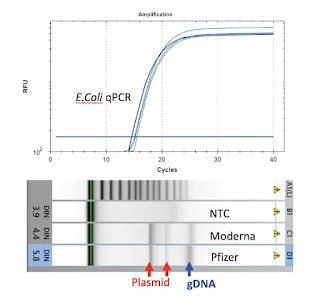
3.2.1. El ADN plasmídico contenido en las vacunas de ARNm es competente para propagarse en células bacterianas En el primer experimento, se determinó si el ADN plasmídico cuya presencia se había inferido a partir de los resultados de secuenciación anteriores es efectivamente biológicamente funcional, en la medida en que puede introducirse en células bacterianas y persistir dentro de ellas. Para ello, se extrajeron de nuevo ácidos nucleicos de las muestras de vacuna. Estos ácidos nucleicos se mezclaron con una suspensión de células de E. coli que se habían hecho competentes para la captación de ADN.
Después de inducir a estas células a captar el ADN y darles algún tiempo para recuperarse, se extendieron sobre placas de Petri llenas de medio de crecimiento solidificado que contenía kanamicina. Como ya se ha señalado, la kanamicina mata cualquier célula de E. coli que no contenga un gen de resistencia a ella. Por lo tanto, el crecimiento observado de colonias bacterianas en esas placas de Petri confirmó que algunas células habían adquirido resistencia a la kanamicina al absorber y propagar los plásmidos. Esto se observó tanto en las muestras de vacuna de Pfizer como en las de Moderna.
En este contexto, cabe señalar que sólo las moléculas de plásmidos circulares, pero no las linealizadas, pueden introducirse eficazmente en las células bacterianas. Por lo tanto, el éxito de este experimento sugiere que algunas de las moléculas de plásmido escaparon a la etapa de linealización (etapa 4 de la sección 1.2) y llegaron hasta el final del proceso de producción en la forma circular que existe en las células bacterianas. Por otra parte, dado que el número de colonias bacterianas observadas en este experimento no era elevado, es probable que la mayor parte del ADN se hubiera linealizado. Dado que los peligros biológicos del ADN extraño dentro de nuestro propio cuerpo pueden variar dependiendo de si es lineal o circular, merece la pena destacar la probable presencia de ambas formas en las vacunas. Queda por determinar la proporción exacta de ADN circular y lineal en las mezclas.
3.2.2. La abundancia de ADN contaminante
El segundo gran hallazgo de este estudio es la cuantificación mediante PCR tanto del ADN como del ARNm contenidos en las muestras de vacunas. Como ya sabrán, en una reacción de PCR, un segmento elegido de una secuencia de ácido nucleico se reduplica mediante síntesis enzimática en varios ciclos de reacción sucesivos. A partir del número de ciclos (o duplicaciones) necesarios para alcanzar una determinada concentración umbral, podemos calcular cuántas copias de la secuencia diana estaban presentes al principio.
En estos experimentos, el formato experimental elegido fue la PCR multiplex, es decir, se amplificaron dos secuencias diana en una única mezcla de reacción. Una de estas dianas se encontraba en el gen de la proteína de la espiga, por lo que debía estar presente tanto en las moléculas de ADN del plásmido como en las moléculas de ARNm de la espiga transcritas a partir de ellas. Para incluir las moléculas de ARNm en esta amplificación, la PCR fue precedida de nuevo por la transcripción inversa.
La otra secuencia diana se encontraba dentro del gen de resistencia a la kanamicina, que debería estar presente únicamente en el ADN plasmídico. Comparando el número de ciclos necesarios para que cada una de las dos dianas cruzara el umbral, se determinó que hasta el 35% del ácido nucleico total contenido en las vacunas es de hecho ADN. A título comparativo, la EMA ha estipulado que el ADN no debe representar más del 0,033% del total de ácidos nucleicos.
3.2.3. Determinación de las secuencias de ADN plasmídico
Los plásmidos contenidos originalmente en las vacunas e introducidos después en células bacterianas (véase el apartado 3.2.1) se aislaron de nuevo de esos cultivos bacterianos y se determinaron sus secuencias completas de ADN. Dichas secuencias se facilitaron íntegramente en el primer estudio de McKernan, pero éste indicó que seguía trabajando para corroborar y perfeccionar los datos de secuenciación. Mientras tanto, las características funcionales del ADN plasmídico hallado en las muestras de la vacuna de Pfizer se muestran en la Figura 1. Se discutirán en relación con el primer estudio de McKernan. Se discutirán en relación con la evaluación de riesgos.
3.3. El tercer informe de McKernan
En su hasta ahora último informe, McKernan examinó ocho viales de un lote anterior de la vacuna de Pfizer utilizando el método de PCR cuantitativa descrito anteriormente. El contenido de ADN en este caso era notablemente inferior al de las muestras de vacunas bivalentes, pero seguía superando el límite de la EMA en un factor de 18-70.
Michael Palmer, MD y Jonathan Gilthorpe, PhD
(Fuente: https://doctors4covidethics.org/; visto en https://childrenshealthdefense.eu/)
Suscribirse a:
Enviar comentarios (Atom)





_a.jpg)



_a.jpg)



No hay comentarios:
Publicar un comentario